近日,图书馆VIP物理系汪骞课题组和上海科技大学免疫化学研究所白芳课题组合作,提出了蛋白质变构路径预测的深度学习模型。该成果在线发表于《先进科学》。
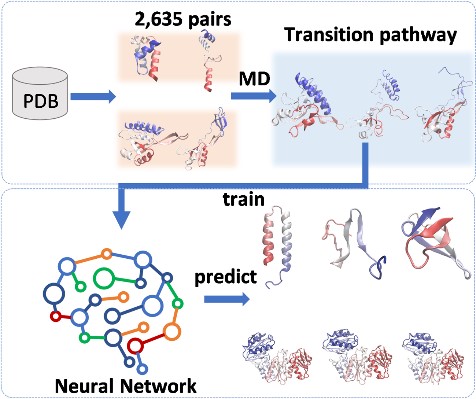
深度学习模型PATHpre建立流程。中国科大供图
近年来,以AlphaFold为代表的深度学习模型在预测蛋白质的静态结构方面取得了巨大成功。然而,蛋白质的功能取决于其动力学特性。研究人员目前正在积极探索其他深度学习算法,旨在预测蛋白质的构象变化等动态行为。开发这类模型的主要挑战之一,在于描述构象转变的动力学数据严重不足。
为了解决该问题,作者结合物理约束的粗粒度分子动力学模型与增强采样方法建立了高效模拟蛋白变构的计算框架,并应用此框架模拟了2635个蛋白质在双稳态之间的构象转变。该工作采集了每个蛋白在其变构路径上的结构信息,从而建立了首个大规模的蛋白质动态数据库。
在此基础上,作者开发了一个通用的深度学习模型PATHpre,能够预测蛋白质在双稳态之间的变构路径。该模型在不同序列长度的蛋白质(范围从44到704个氨基酸)上均展现了良好的预测能力,并适用于包括变形蛋白在内的多类变构体系。研究人员在多个系统中展示了预测结果与实验或模拟数据的一致性,并利用该模型发现了肌球蛋白的变构调控新机制。
相关论文信息:https://onlinelibrary.wiley.com/doi/10.1002/advs.202400884
作者:王敏 来源:中国科学报 发布时间:2024/10/14
原文链接:https://news.sciencenet.cn/htmlnews/2024/10/531529.shtm